La estructura 3D del receptor ACE2 que usa el coronavirus SARS-CoV-2 para infectar
Por Francisco R. Villatoro, el 7 marzo
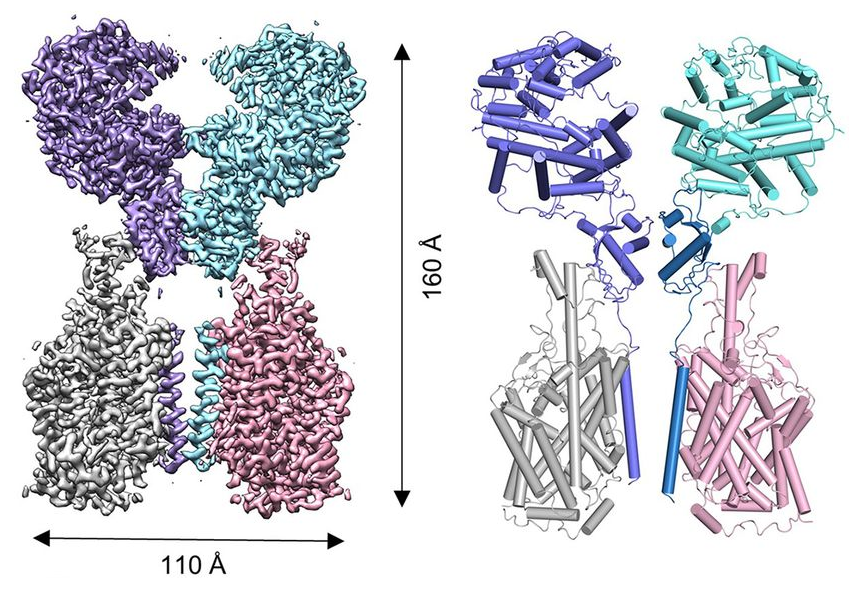
La reconstrucción 3D por criomicroscopia electrónica (cryo-EM) más
esperada tras la de «la glicoproteína espicular del coronavirus
SARS-CoV-2» (LCMF, 24 feb 2020) era la del receptor ACE2 de la célula huésped. Se acaba de publicar en Science para el complejo ACE2-B0AT1 con una resolución de 2.9 Å; el transportador de aminoácidos B0AT1
no interfiere en la interacción entre la enzima convertidora de
angiotensina 2 (ACE2) y el dominio de unión al receptor (RBD) del virus,
pero facilita la preparación criomicroscópica. La reconstrucción de la
unión ACE2-RBD se ha logrado con una resolución local de 3.5 Å, lo que
permite entender multitud de detalles bioquímicos de gran relevancia
biomédica. Se ha dado un paso de gigante en la comprensión de las bases
moleculares del reconocimiento y la infección por el coronavirus de
Wuhan.
La función biológica de la proteína ACE2 es la maduración de la
angiotensina, una hormona que controla la vasoconstricción y la presión
arterial. ACE2 es una proteína de membrana que se expresa en pulmones,
el corazón, los riñones y el intestino. Algunos coronavirus aprovechan
esta proteína para su infección, entre ellos, SARS-CoV-2, que causa la
enfermedad por coronavirus 2019 (COVID-19) y SARS-CoV, que causa el
SARS, con quien comparte un 80% de su ARN —recuerda que comparte un 96%
con el coronavirus de murciélago BatCoV RaTG13—. La glicoproteína
espicular (proteína S) del virus es una proteína trimérica que se
escinde en dos subunidades (S1 y S2) durante la infección. En el dominio
S1 se encuentra la región que se une al virus, mientras que S2 es
responsable de la fusión de las membranas (más detalles en LCMF, 24 feb 2020).
La nueva imagen por cryo-EM de la unión entre las proteínas S y ACE2
ayudará al desarrollo vacunas y antivirales. Sin lugar a dudas se está
avanzando en el conocimiento molecular de la infección por SARS-CoV-2 a
un ritmo de vértigo. El artículo es Renhong Yan, Yuanyuan Zhang, …,
Qiang Zhou, «Structural basis for the recognition of the SARS-CoV-2 by
full-length human ACE2,» Science 367: 1444-1448 ( 27 Mar 2020), doi: https://doi.org/10.1126/science.abb2762.

Sobre la microscopia crioelectrónica, más conocida como
criomicroscopia electrónica, o cryo-EM, premio Nobel de Química 2017,
puedes consultar LCMF, 05 oct 2017.
La idea es distribuir un gran número de biomoléculas sobre un sustrato
en diferentes orientaciones y analizar las imágenes de un microscopio
electrónico para obtener una reconstrucción de su estructura
tridimensional. En este nuevo trabajo se ha reconstruido el complejo
ACE2-B0AT1 en lugar de solo la proteína ACE2, porque así se
facilita la preparación de la muestra para el microscopio electrónico.
Como B0AT1 es una proteína de membrana no afecta a la unión
entre ACE2 y el receptor RBD del coronavirus. La reconstrucción 3D de la
estructura con una resolución de 2.9 Å se ha obtenido a partir de 418
140 moléculas del complejo sobre el sustrato; en algunos dominios
extracelulares se ha alcanzado 2.7 Å, mientras que en la unión con el
receptor RBD solo se ha logrado unos 3.5 Å.
El complejo es un dímero y en la figura ha sido coloreado por
subunidades. El dominio de proteasa (PD) en celeste y el dominio similar
a la colectrina (CLD) en azul —la colectrina también se llama TMEM27
(proteína transmembrana 27)—. La ACE2 tiene dos conformaciones,
«cerrada» cuando las dos PD están en contacto entre sí, y «abierta»,
cuando están separadas (en la figura se muestra la conformación
«cerrada»). Los interesados en más detalles sobre el papel de los
diferentes dominios en esta proteína pueden consultar el artículo
científico, que es de acceso gratuito (open access).

Lo más relevante del nuevo artículo es la reconstrucción 3D del complejo RBD-ACE2-B0AT1
que muestra en gran detalle la unión entre ACE2 de la célula huésped y
RBD del coronavirus. En este complejo la proteína ACE2 está en
conformación «cerrada» (no se observó en ningún caso en la «abierta») y
se usaron 527,017 biomoléculas del complejo sobre el sustrato para
lograr una resolución de 3.5 Å para RBD. Cada PD se une a sendas RBD del
SARS-CoV-2 en una configuración muy similar a la que existe entre ACE2 y
el SARS-CoV.

La unión entre ACE2 y RBD se puede dividir en tres grupos: el extremo
N (amino) de la hélice α1, un puente intermedio entre extremos N y C
(carboxilo) de la hélice α1 con participación de la α2, y un bucle en
las láminas β3 y β4. Los interesados en el papel concreto de cada uno de
los aminoácidos involucrados en esta unión pueden consultar el artículo
en Science. Mi objetivo con esta pieza es solo ilustrar el gran lujo de detalles que se ha desvelado sobre la unión ACE2-RBD.
-
Esta figura ilustra una comparación (superposición de imágenes) entre
la unión ACE2-RBD para los coronavirus SARS-CoV-2 (amarillo) y SARS-CoV
(verde). A pesar de la semejanza general, se observan diferencias en la
secuencia de aminoácidos en las regiones de unión; recuerda que los
aminóacidos se denotan por una letra mayúscula y un número que indica su
posición en la secuencia (de la proteína S en este caso); por ejemplo,
F456 es una fenilalanina (Phe) en la posición 456 de la proteína S de
SARS-CoV-2, mientras L443 es una leucina (Leu) en la posición 443 de la
de SARS-CoV. El conocimiento detallado de estos residuos permitirá saber
qué fármacos antivirales contra SARS pueden ser útiles contra COVID-19 y
ayudará a diseñar otros más eficaces.
El conocimiento detallado de la estructura tridimensional de las
proteínas en interacción ayuda a comprender su función bioquímica. Por
supuesto, la estructura tridimensional del complejo RBD-ACE2-B0AT1
es solo el primer paso para entender todos los detalles del proceso de
reconocimiento entre el coronavirus y la célula huésped, y la posterior
fusión de sus membranas para permitir la entrada del ARN vírico. La
criomicroscopia electrónica está revolucionando nuestro conocimiento
estructural en biología molecular.
.https://francis.naukas.com/2020/03/07/la-estructura-3d-del-receptor-ace2-que-usa-el-coronavirus-sars-cov-2-para-infectar/?fbclid=IwAR2TY__x4mPdGW47v7SOlIvTfnGMdqZgcw03quOogZj3CPptBXobuoDCt8I
No hay comentarios:
Publicar un comentario