“La información guardada en nuestros cromosomas forma un manual de instrucciones para todas las células de nuestro cuerpo. Cada célula contiene los mismos cromosomas, por tanto tiene la misma información. ¿Qué permite a una célula diferenciarse entre un músculo o un nervio?”
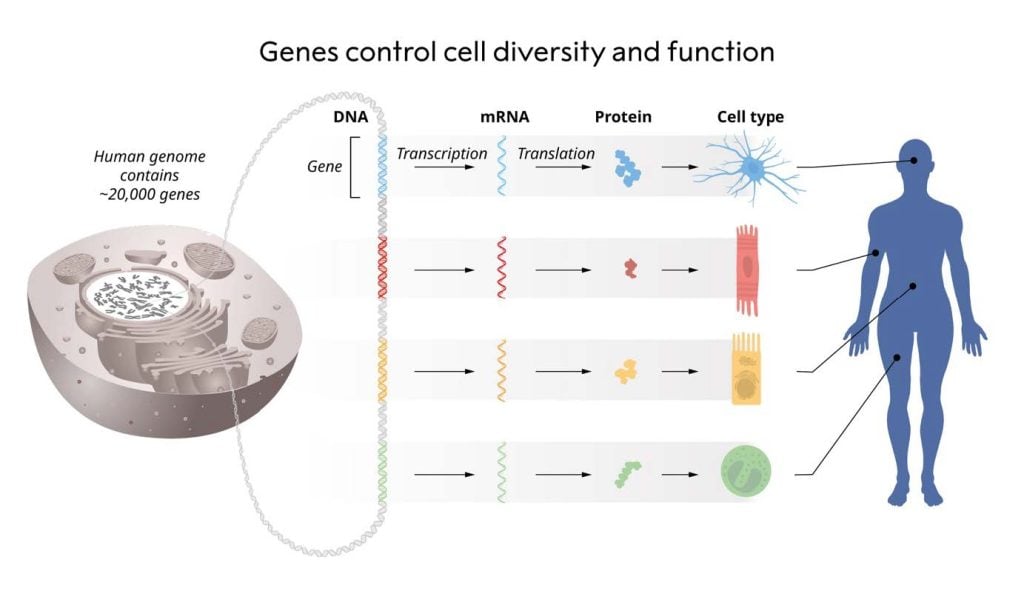
El Premio Nobel en Fisiología y/o Medicina de este año se enfoca en el descubrimiento de un mecanismo regulador vital utilizado en las células para controlar la actividad genética. La información genética pasa del ADN al ARN mensajero (ARNm), a través de un proceso llamado transcripción, y luego a la maquinaria celular para la producción de proteínas. Allí, el ARNm se traduce para que las proteínas se produzcan de acuerdo con las instrucciones genéticas almacenadas en el ADN, que sería como un libro de recetas. Desde mediados del siglo XX, varios de los descubrimientos científicos han explicado cómo funcionan estos procesos. Nuestros órganos y tejidos constan de muchos tipos de células diferentes, todas con información genética idéntica almacenada en su ADN.
Sin embargo, estas diferentes células expresan conjuntos únicos de proteínas. Pero ¿cómo es esto posible?
La respuesta está en la regulación precisa de la actividad genética, de modo que sólo el conjunto correcto de genes esté activo en cada tipo de célula específico. Esto permite, por ejemplo, que las células musculares, las células intestinales y diferentes tipos de células nerviosas realicen funciones especializadas. Además, la actividad genética debe ajustarse continuamente para adaptar las funciones celulares a las condiciones cambiantes de nuestro cuerpo y nuestro entorno. Si la regulación genética falla, puede provocar enfermedades graves como cáncer, diabetes o autoinmunidad. Por esto, comprender la regulación de la actividad genética ha sido un objetivo importante durante muchas décadas. En la década de 1960, se demostró que proteínas especializadas, conocidas como factores de transcripción, pueden unirse a regiones específicas del ADN y controlar el flujo de información genética determinando qué ARNm se produce. Desde entonces se han identificado miles de factores de transcripción y durante mucho tiempo se creyó que se habían resuelto los principios fundamentales de la regulación genética. Sin embargo, en 1993, los premios Nobel de este año publicaron hallazgos inesperados que describían un nuevo nivel de regulación genética, que resultó ser muy significativo y se conserva a lo largo de la evolución
A finales de la década de 1980, Victor Ambros y Gary Ruvkun becarios postdoctorales en el laboratorio de Robert Horvitz - quien recibió el Premio Nobel en 2002, junto con Sydney Brenner y John Sulston. En el laboratorio de Horvitz, estudiaron un gusano redondo relativamente sencillo de 1 mm de largo, C. elegans. A pesar de su tamaño, C. elegans posee muchos tipos de células especializadas, como células nerviosas y musculares, que también se encuentran en animales más grandes y complejos, lo que lo convierte en un modelo útil para investigar cómo se desarrollan y maduran los tejidos en organismos multicelulares. Ambros y Ruvkun estaban interesados en genes que controlan el momento de activación de diferentes programas genéticos, que aseguran que varios tipos de células se desarrollen en el momento preciso
Estudiaron dos cepas mutantes de gusanos, lin-4 y lin-14, que mostraban defectos en el momento de activación de programas genéticos durante el desarrollo. Ellos querían identificar los genes mutados y comprender su función. Ambros había demostrado previamente que el gen lin-4 parecía ser un regulador negativo del gen lin-14. Sin embargo, se desconocía cómo se bloqueaba la actividad de lin-14. Ambros y Ruvkun estaban intrigados por estos mutantes y su posible relación, y ellos se propusieron resolver este misterio. Victor Ambros estudió el gen mutado lin-4 en su laboratorio. Un mapeo metódico permitió la clonación del gen y condujo a un hallazgo inesperado. El gen lin-4 produjo una molécula de ARN inusualmente corta que carecía de un código para la producción de proteínas. Estos sorprendentes resultados sugirieron que este gen pequeñísimo era responsable de inhibir lin-14. ¿Cómo podría funcionar esto? Al mismo tiempo, Gary Ruvkun investigó la regulación del gen lin-14. A diferencia de lo que se conocía entonces en cómo funcionaba la regulación genética, Ruvkun demostró que no es la producción de ARNm a partir de lin-14 lo que inhibe lin-4. La regulación parecía ocurrir en una etapa posterior del proceso de expresión genética, mediante la interrupción de la producción de proteínas. Los experimentos también revelaron un segmento en el ARNm de lin-14 que era necesario para su inhibición por lin-4. Los dos galardonados compararon sus hallazgos y dieron como resultado un descubrimiento revolucionario. La secuencia corta de lin-4 coincidía con secuencias complementarias en el segmento crítico del ARNm de lin-14. Ambros y Ruvkun
Realizaron más experimentos que demostraron que el microARN lin-4 desactiva a lin-14 al unirse a las secuencias complementarias en su ARNm, bloqueando la producción de la proteína lin-14. ¡Se había descubierto un nuevo principio de regulación genética, mediado por un tipo de ARN
Los resultados fueron publicados en 1993 en dos artículos en la revista Cell, que fueron recibidos con un silencio casi ensordecedor por parte de la comunidad científica. Aunque eran interesantes, el inusual mecanismo de regulación genética se consideró una peculiaridad de C. elegans, probablemente irrelevante para humanos y animales más complejos. Esa percepción cambió en 2000, cuando Ruvkun publicó su descubrimiento de otro microARN, que a diferencia de lin-4, estaba muy conservado y presente en todo el reino animal el gen let-7.
Esta vez el artículo despertó gran interés y, durante los años siguientes se identificaron cientos de microARN diferentes. Hoy en día sabemos que existen más de mil genes para diferentes microARN en humanos y que la regulación genética por microARN es universal entre organismos
La unión de microARN conduce a la inhibición de la síntesis de proteínas o a la degradación del ARNm. Curiosamente, un solo microARN puede regular la expresión de muchos genes diferentes y, a la inversa, un solo gen puede ser regulado por múltiples microARN, coordinando y ajustando así redes enteras de genes.
La regulación genética mediante microARN, revelada por vez primera por Ambros y Ruvkun, ha estado funcionando durante cientos de millones de años. Este mecanismo ha permitido la evolución de organismos cada vez más complejos. Sabemos por investigaciones genéticas que las células y los tejidos no se desarrollan normalmente sin microARN. Una regulación anormal por microARN puede contribuir al cáncer, y se han encontrado mutaciones en genes que codifican microARN en humanos, causando afecciones como pérdida auditiva congénita y trastornos oculares y esquéleticos. Este descubrimiento fundamental de Ambros y Ruvkun en un pequeño gusano C. elegans fue inesperado, revelando una nueva dimensión de la regulación genética, esencial para todas las formas de vida complejas.
- https://x.com/chayito09/status/1843343080897864098
Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans
https://www.cell.com/fulltext/0092-8674(93)90530-4
The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14
https://www.cell.com/fulltext/0092-8674%2893%2990529-Y
Premio Nobel de Medicina 2024 para los descubridores del microARN y su papel en la regulación genética

El Instituto Karolinska de Suecia ha otorgado el premio Nobel en Fisiología o Medicina de 2024 a dos científicos estadounidenses por su descubrimiento de un principio fundamental que rige la regulación de la actividad genética. El comité de los Nobel reconoce el trabajo de Victor Ambros y Gary Ruvkun por el descubrimiento del microARN , una nueva clase de pequeñas moléculas de ARN que son esenciales para todos los organismos vivos.
Los dos científicos estadounidenses descubrieron los microARN, una nueva clase de diminutas moléculas de ARN que desempeñan un papel crucial en la regulación de los genes.
Su revolucionario descubrimiento en el pequeño gusano C. elegans reveló un principio completamente nuevo de regulación genética, que resultó ser esencial para los organismos pluricelulares, incluido el ser humano.
Durante décadas, la comunidad científica prestó poca atención a este descubrimiento. Según la Fundación Nobel, se pensaba que este mecanismo era "una peculiaridad" de un gusano, algo "probablemente irrelevante para los humanos y otros animales más complejos". Pero no.
De hecho, "ahora se sabe que el genoma humano codifica más de 1.000 microARN", explica la Fundación Nobel.
Los ganadores se repartirán un premio de 11 millones de coronas suecas (aproximadamente US$1 millón).
Particularidades genéticas
Todas las células del cuerpo humano contienen la misma información genética en bruto, encerrada en nuestro ADN.
Pero, a pesar de partir de la misma información genética, las células del cuerpo humano son muy diferentes en forma y función.
Los impulsos eléctricos de las células nerviosas son distintos de los latidos rítmicos de las células cardíacas. La central metabólica que es una célula hepática es distinta de una célula renal que filtra la urea de la sangre. La capacidad de detección de la luz de las células de la retina es distinta de la de los glóbulos blancos que producen anticuerpos para combatir las infecciones.
Toda esta variedad puede surgir del mismo material de partida gracias a la expresión genética.
Los científicos estadounidenses fueron los primeros en descubrir los microARN y cómo estos ejercen un control sobre la forma en que los genes se expresan de manera diferente en los distintos tejidos.
Sin la capacidad de controlar la expresión génica, cada célula de un organismo sería idéntica, por lo que los microARN ayudaron a posibilitar la evolución de formas de vida complejas.
La regulación anómala de los microARN puede contribuir al cáncer y a algunas enfermedades como la pérdida de audición congénita y los trastornos óseos.
Un ejemplo es el síndrome DICER1, que provoca cáncer en diversos tejidos y está causado por mutaciones que afectan a los microARN.
Cómo funciona
Ambros y Ruvkun investigaron sobre el gusano nematodo C. elegans.
"Vieron unos gusanos que parecían un poco raros y decidieron entender por qué", cuenta Olle Kämpe, miembro del Comité Nobel.
Experimentaron con una forma mutante del gusano que no desarrollaba algunos tipos de células.
Y finalmente dieron con pequeños fragmentos de material genético o microARN que eran esenciales para el desarrollo de los gusanos.
Así es como funciona:
- Nuestro ADN contiene un gen o instrucción genética.
- Nuestras células hacen una copia, llamada ARN mensajero o simplemente ARNm (lo recordarás de las vacunas covid).
- Este ARN sale del núcleo de la célula y ordena a las fábricas de proteínas de la célula que empiecen a producir una proteína específica.
- Pero los microARN se interponen en el camino adhiriéndose al ARN mensajero y detienen su funcionamiento.
- En esencia, el mircoARN ha impedido que el gen se exprese en la célula.
Otros trabajos demostraron que no se trata de un proceso exclusivo de los gusanos, sino de un componente esencial de la vida en la Tierra.
Fuente de la imagen, Getty Images
"Su trabajo pionero sobre la regulación genética por microARN allanó el camino a investigaciones sobre terapias novedosas para enfermedades devastadoras como la epilepsia, pero también nos abrieron los ojos a la maravillosa maquinaria que controla estrechamente lo que ocurre en nuestras células", dijo a la BBC Janosch Heller de la Universidad de la Ciudad de Dublín.
Los ganadores de los premios de Medicina y Fisiología son elegidos por la Asamblea Nobel del Instituto Karolinska de Suecia.
El Premio Nobel de Fisiología o Medicina se ha concedido 114 veces a 227 galardonados entre 1901 y 2023.
El año pasado, el premio fue otorgado a Katalin Karikó y Drew Weissman por la vacuna de la covid basada en ARN mensajero.
Premio Nobel de Medicina 2023 por las vacunas de ARN contra la covid-19
La investigación reconocida con el galardón este año se centra en el descubrimiento de un mecanismo regulador vital que se utiliza en las células para controlar la actividad genética. La información genética fluye del ADN al ARN mensajero (ARNm), a través de un proceso llamado transcripción, y luego a la maquinaria celular para la producción de proteínas. Allí, los ARNm se traducen para que las proteínas se fabriquen de acuerdo con las instrucciones genéticas almacenadas en el ADN.
Victor Ambros y Gary Ruvkun (nacidos en New Hampshire en 1953 y en Berkeley, California, en 1952, respectivamente) se interesaron por el modo en que se desarrollan los distintos tipos de células y revelaron un principio completamente nuevo de regulación genética que resultó ser esencial para los organismos multicelulares, incluidos los humanos. Ahora se sabe que el genoma humano codifica más de mil microARN.
Un nuevo nivel de regulación genética
Desde mediados del siglo XX, varios de los descubrimientos científicos más fundamentales han explicado cómo funcionan estos procesos. Nuestros órganos y tejidos están compuestos por muchos tipos de células diferentes, todas con información genética idéntica almacenada en su ADN. Sin embargo, estas diferentes células expresan conjuntos únicos de proteínas. ¿Cómo es esto posible?
En la década de 1960 se demostró que unas proteínas especializadas, conocidas como factores de transcripción, pueden unirse a regiones específicas del ADN y controlar el flujo de información genética al determinar qué ARNm se producen. Desde entonces, se han identificado miles de factores de transcripción y durante mucho tiempo se creyó que se habían resuelto los principios básicos de la regulación genética. Sin embargo, en 1993, los premios Nobel de ese año publicaron hallazgos inesperados que describían un nuevo nivel de regulación genética, que resultó ser muy significativo y se mantuvo a lo largo de la evolución.
El gusanito que lo cambió todo
A finales de los años 1980, Victor Ambros y Gary Ruvkun fueron becarios postdoctorales en el laboratorio de Robert Horvitz, donde estudiaron un gusano redondo relativamente modesto de 1 mm de largo, C. elegans. A pesar de su pequeño tamaño, C. elegans posee muchos tipos de células especializadas, como células nerviosas y musculares que también se encuentran en animales más grandes y complejos, lo que lo convierte en un modelo útil para investigar cómo se desarrollan y maduran los tejidos en organismos multicelulares.
Ambros y Ruvkun estaban interesados en los genes que controlan el momento de activación de diferentes programas genéticos, asegurando que varios tipos de células se desarrollen en el momento adecuado. Estudiaron dos cepas mutantes de gusanos, lin-4 y lin-14, que mostraban defectos en el momento de activación de los programas genéticos durante el desarrollo. Los galardonados querían identificar los genes mutados y comprender su función lo que les llevó a descubrir un nuevo principio de regulación genética, mediado por un tipo de ARN previamente desconocido, el microARN.
Los resultados fueron recibidos inicialmente con un silencio casi ensordecedor por parte de la comunidad científica
Los resultados fueron publicados en 1993 en dos artículos en la revista Cell y fueron recibidos inicialmente con un silencio casi ensordecedor por parte de la comunidad científica. Aunque eran interesantes, el inusual mecanismo de regulación genética se consideró una peculiaridad de C. elegans , probablemente irrelevante para los humanos y otros animales más complejos. Esa percepción cambió en 2000 cuando el grupo de investigación de Ruvkun publicó su descubrimiento de otro microARN que estaba altamente conservado y presente en todo el reino animal. El artículo despertó un gran interés y, en los años siguientes, se identificaron cientos de microARN diferentes. Hoy, sabemos que hay más de mil genes para diferentes microARN en humanos, y que la regulación genética por microARN es universal entre los organismos multicelulares.
La regulación genética por microARN ha estado en funcionamiento durante cientos de millones de años y ha permitido la evolución de organismos cada vez más complejos. Sabemos por la investigación genética que las células y los tejidos no se desarrollan normalmente sin microARN. La regulación anormal por microARN puede contribuir al cáncer, y se han encontrado mutaciones en los genes que codifican microARN en humanos, causando afecciones como pérdida de audición congénita y trastornos oculares y esqueléticos. Las mutaciones en una de las proteínas necesarias para la producción de microARN dan lugar al síndrome DICER1, un síndrome raro pero grave vinculado al cáncer en varios órganos y tejidos.
Investigación en enfermedades
“Aunque los genes de microARN son muy pequeños (de 21 a 25 nucleótidos), existen enfermedades genéticas hereditarias causadas por mutaciones en estos genes y, como consecuencia, se desregulan muchos genes distintos, pudiendo alterar varias vías de señalización o metabólicas y afectar a distintos órganos”, señala Gemma Marfany, catedrática de Genética de la Universidad de Barcelona (UB), al SMC.
“La denominación de microARN proviene de que son unas secuencias muy pequeñas, de tan solo 22 nucleótidos, pero su potencial en la célula es enorme”, añade Guillermo Peris Ripollés, profesor titular de la Universitat Jaume I cuyo grupo investiga la desregulación de los microARN. “De hecho, aumentos o disminuciones de su expresión están asociados a distintas enfermedades”.
Sònia Guil, líder del Grupo de Regulación del ARN y Cromatina del Instituto de Investigación contra la Leucemia Josep Carreras, destaca especialmente que el descubrimiento de estos pequeños RNAs tuvo lugar en unos gusanos diminuto. “Reconocer el trabajo de estos investigadores es poner de relieve lo esencial de la investigación básica (incluyendo organismos modelos no humanos), lo cual muchas veces es poco valorado por las decisiones en política científica”, indica, también al SMC.
-------
El Nobel que han anunciado hoy, intenta responder a una pregunta básica: Si todas las células tienen el mismo ADN, ¿cómo hacen para diferenciarse unas de otras?
¿Por qué unas células se convierten en neuronas y otras en músculo y otras en intestino, si todas tienen el mismo ADN?
For the discovery of microRNA and its role in post-transcriptional gene regulation
La evolución de los organismos pluricelulares a partir de ancestros unicelulares, en los que cada tipo celular adquirió funciones especializadas, requirió mecanismos cada vez más sofisticados de regulación génica. Además de la regulación génica transcripcional mediada por factores de unión al ADN que actúan sobre secuencias reguladoras, surgieron otras formas de sistemas de control a medida que evolucionaban organismos cada vez más complejos. A lo largo de cientos de millones de años, los genes que codifican diminutas moléculas de ARN no codificante, los llamados microARN, se expandieron dentro de los genomas de los organismos multicelulares para ejercer un control postranscripcional sobre la estabilidad del ARNm y la traducción de proteínas. Los microARN y su modo de regulación génica permanecieron completamente desconocidos hasta el descubrimiento de Victor Ambros y Gary Ruvkun en 1993. Los dos premios Nobel investigaron nematodos mutantes de C. elegans con defectos de desarrollo causados por alteraciones en los loci genéticos lin-4 y lin-14. El laboratorio de Ambros clonó el gen lin-4 e hizo el sorprendente descubrimiento de que no codificaba una proteína. En su lugar, codificaba un ARN corto no codificante de 22 nucleótidos. Paralelamente, el laboratorio de Ruvkun determinó que lin-4 regula lin-14 a través de elementos multipl en su región 3′ no traducida (3'UTR). Al comparar la información de secuencias, definieron una complementariedad parcial de secuencias entre el ARN corto no codificante de lin-4 y los elementos 3'UTR de lin-14. Esto proporcionó una primera visión de un tipo conceptualmente novedoso de ARN reguladores: los microARN. En 2000, el laboratorio de Ruvkun descubrió el microARN let-7, altamente conservado, lo que llevó a la posterior identificación de microARN homólogos en diversas especies animales, incluida la humana. Esto desencadenó intensos esfuerzos de clonación y secuenciación para identificar microARNs en todo el reino animal, lo que condujo al descubrimiento de que los microARNs abarcan un gran grupo de reguladores que controlan vastas redes de genes codificadores de proteínas. El descubrimiento de Ambros y Ruvkun fue totalmente inesperado y desveló un mecanismo regulador postranscripcional evolutivamente conservado mediado por microARN, con funciones críticas en el desarrollo animal y en la función de los tejidos adultos.
Introduction
El control de cuándo y dónde debe transcribirse cada gen en ARN y traducirse en proteína es un aspecto fundamental de la vida (Figura 1). Por ejemplo, la insulina se produce en las células beta de los islotes pancreáticos, mientras que las opsinas se producen en la retina del ojo. Las instrucciones para la regulación precisa de los genes específicos de cada tipo celular están codificadas en el propio material genético y actúan sobre él proteínas de unión al ADN específicas de cada secuencia. François Jacob y Jacque Monod recibieron el Premio Nobel de Fisiología o Medicina en 1965 por descubrir cómo se regulan los genes. El repertorio de factores de transcripción de unión al ADN está bien conservado entre los eucariotas unicelulares y pluricelulares (King et al., 2008), mientras que en los organismos pluricelulares han surgido capas adicionales de regulación génica para garantizar la producción correcta de ARN y proteínas en un momento dado en cada tipo celular.
En la década de 1970, las pruebas de mutagénesis en C. elegans realizadas en el laboratorio de Brenner descubrieron el mutante lin-4 (e912). Estos gusanos mostraban un fenotipo sorprendente: muchos tipos celulares y estructuras morfológicas estaban totalmente ausentes, y los huevos se acumulaban debido a un fallo en el desarrollo de la vulva (Figura 2) (Horvitz y Sulston, 1980; Chalfie, Horvitz y Sulston, 1981), aparentemente por la reiteración de programas de desarrollo para linajes celulares específicos.
La importante alteración del desarrollo del gusano observada en el mutante lin-4 sugería que lin-4 codificaba un regulador maestro del calendario de desarrollo. Se caracterizó un gran número de mutantes heterocrónicos adicionales que presentaban diversos defectos temporales del desarrollo, incluido un segundo mutante, lin-14, descubierto en el laboratorio de Horvitz (Ferguson, Sternberg y Horvitz, 1987).
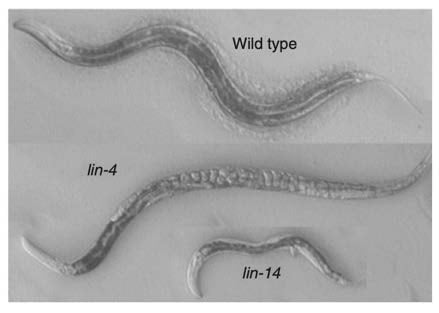 Figure 2. Heterochronic worm mutants with developmental defects. Nematode lin-4 and lin-14 mutants with disrupted animal development. Mutant lin-4 worms reiterate developmental programs for cell lineages to accumulate internal eggs without forming a vulva, while lin-14 mutants are small and lack larval development. Worms adapted from (Ambros, 2008)
Figure 2. Heterochronic worm mutants with developmental defects. Nematode lin-4 and lin-14 mutants with disrupted animal development. Mutant lin-4 worms reiterate developmental programs for cell lineages to accumulate internal eggs without forming a vulva, while lin-14 mutants are small and lack larval development. Worms adapted from (Ambros, 2008)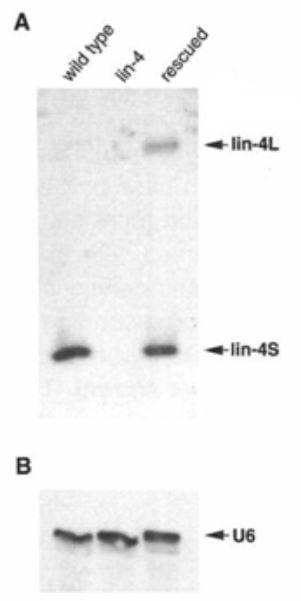 Figure 3. Identification of two short lin-4 transcripts. Northern blot of total RNA from wild-type, lin-4 (e912) mutant, and lin-4 (e912) mutant rescued with Sal I fragment, probed with radiolabeled lin-4 RNA probe, compared to U6 loading control. (Lee, Feinbaum and Ambros, 1993).
Figure 3. Identification of two short lin-4 transcripts. Northern blot of total RNA from wild-type, lin-4 (e912) mutant, and lin-4 (e912) mutant rescued with Sal I fragment, probed with radiolabeled lin-4 RNA probe, compared to U6 loading control. (Lee, Feinbaum and Ambros, 1993).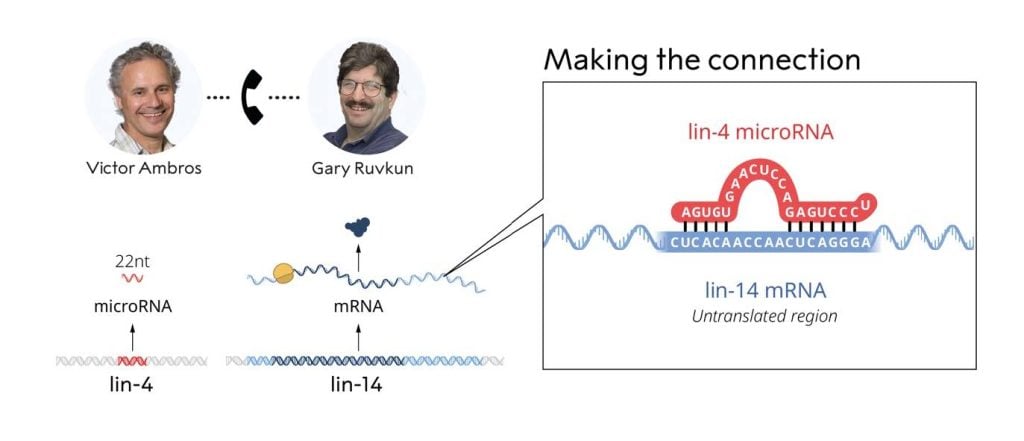 Figure 4. Complementary sequence elements in lin-4 and lin-14 RNA. Upon comparing cloned sequences for lin-4 and lin-14, it was revealed that the short 22 nt lin-4 RNA had partial complementarity to repeated elements in the lin-14 3’UTR. © The Nobel Committee for Physiology or Medicine. Ill. Mattias Karlén
Figure 4. Complementary sequence elements in lin-4 and lin-14 RNA. Upon comparing cloned sequences for lin-4 and lin-14, it was revealed that the short 22 nt lin-4 RNA had partial complementarity to repeated elements in the lin-14 3’UTR. © The Nobel Committee for Physiology or Medicine. Ill. Mattias Karlén Figure 5. Evolutionary conservation of the let-7 RNA expression and microRNAs more generally.
Left: An evolutionary tree of metazoans, highlighting the branches of
the tree with a detectable let-7 microRNA expression (+) or where no let-7
expression was detected (-). Species with similar developmental pattern
of let-7 RNA expression is (no let-7 in early stages; but let-7
expression by adulthood) are indicated by ‘Dev.’. (Pasquinelli et al.,
2000). Right: MicroRNA genes have evolved and expanded within the
genomes of multicellular organisms for over 500 million years. © The Nobel Committee for Physiology or Medicine. Ill. Mattias Karlén
Figure 5. Evolutionary conservation of the let-7 RNA expression and microRNAs more generally.
Left: An evolutionary tree of metazoans, highlighting the branches of
the tree with a detectable let-7 microRNA expression (+) or where no let-7
expression was detected (-). Species with similar developmental pattern
of let-7 RNA expression is (no let-7 in early stages; but let-7
expression by adulthood) are indicated by ‘Dev.’. (Pasquinelli et al.,
2000). Right: MicroRNA genes have evolved and expanded within the
genomes of multicellular organisms for over 500 million years. © The Nobel Committee for Physiology or Medicine. Ill. Mattias KarlénFollowing the discovery of let-7, several research labs sought to identify additional microRNAs in humans and other species via small RNA cloning. The laboratory of Thomas Tuschl cloned novel microRNAs from human and fruit fly tissues (Lagos-Quintana et al., 2001), David Bartel’s lab isolated new microRNAs from the nematode (Lau et al., 2001), as did Ambros lab (Lee and Ambros, 2001). The collective evidence was now compelling: a vast class of regulatory microRNA exists across animals, likely playing important roles in gene regulation. Advances in molecular biology and sequencing technologies have since led to the identification of over a thousand microRNA genes in the human genome. Currently, miRBase, a database for microRNA genes, comprises over 38,000 hairpin precursors and 48,860 mature microRNA gene sequences across 271 organisms (Kozomara, Birgaoanu and Griffiths-Jones, 2019). Even viruses have been found to encode microRNA genes (Pfeffer et al., 2004).
The cloning of additional microRNAs and the availability of whole genome sequences presented increasing opportunities to define the base-pairing rules between microRNAs and 3’UTR regions. Pivotal studies conducted in the laboratories of David Bartel, Christopher Burge and Stephen Cohen (Lewis et al., 2003; Stark et al., 2003; Brennecke et al., 2005; Lewis, Burge and Bartel, 2005) elucidated the overall rules of microRNA target recognition using combined experimental and comparative genomics approaches. These studies showed that microRNAs typically have partial complementarity to the target mRNAs, primarily in the microRNA “seed” region. This work also unveiled that each microRNA likely regulat multiple protein-coding genes, as many 3’UTRs exhibit excessive conservation of sequences complementary to microRNA seed sequences (Brennecke et al., 2005; Lewis, Burge and Bartel, 2005). Interestingly, genes coexpressed with a cell-type or lineage specific microRNA are devoid of target sites for that specific microRNA. In contrast, such microRNA target sites are common in genes expressed in neighboring cells and tissues (Farh et al., 2005; Stark et al., 2005). These observations reinforced the hypothesis that microRNAs have important functions in cell lineage formation and cell-type stability in multicellular organisms.
MicroRNA biogenesis and function
Parallel to the cloning of additional microRNA genes, intense efforts by several research groups were devoted to understanding microRNA biogenesis and mechanisms of action (Bartel, 2004). Strategies for microRNA gene transcription vary. Many microRNA genes are independent transcriptional units, sometimes clustered, while others reside within introns of protein-encoding genes. Canonical primary microRNAs (pri-microRNAs) are transcribed by RNA polymerase II and feature a hairpin-structured sequence. This hairpin serves as a substrate for processing in the nucleus by the microprocessor, a heterotrimeric complex containing Drosha endonuclease, to cleave both strands to produce the precursor microRNA (pre-microRNA), typically 60-70 nucleotides long, first detected in the Ambros lab (Figure 2). Exportin 5 and RAN-GTP facilitate pre-microRNA transport to the cytoplasm. Subsequent processing by Dicer, an endonuclease originally identified in Greg Hannon’s laboratory (Bernstein et al., 2001), forms a microRNA duplex. The effective microRNA strand becomes loaded onto an Argonaute protein-containing silencing complex, whereas the other “passenger” strand is displaced (Schwarz et al., 2003). Once the microRNA strand is loaded into the silencing complex, it can carry out sequence-specific negative regulation of mRNAs via reduced translation and/or mRNA degradation. This regulation involves the adaptor protein TNRC6 and the poly(A)-binding protein PABPC that recruit deadenylase complexes that shorten the mRNA polyA tail, resulting in mRNA degradation and translational inhibition depending on the cellular context, e.g. developmental stage and cell type.
The machinery that processes and executes microRNA function is also used for other RNA-based silencing mechanisms, commonly known as RNA interference (RNAi). These include small interfering RNAs (siRNAs), endogenous piwi-associated RNAs (piRNAs) and repeat-associated small interfering RNA (rasiRNAs). The discovery that double-stranded RNA can induce sequence-dependent gene silencing (Fire et al., 1998), earned Andrew Z. Fire and Craig C. Mello the 2006 Nobel Prize in Physiology or Medicine. Whereas RNAi functions primarily as a defense mechanism against virus infections (in plants and in animals of lower complexity) and against unwanted genomic mobile element activity, microRNAs exert post-transcriptional control over mRNAs throughout development and across adult cell types. To this end, microRNAs have evolved partial complementarity towards their target mRNA sequences to “tune” the respective effects on each mRNA target, whereas e.g. siRNAs are often exogenous with complete complementarity to specific RNA target sequences that become cleaved. In 1999, David Baulcombe showed that post-transcriptional gene silencing in plants involves processing of short RNAs with specificity to target sequences (Hamilton and Baulcombe, 1999), further connecting observations in different fields.
Evolution of microRNAs and their physiological roles
La aparición y expansión de genes de microARN están íntimamente ligadas a la evolución de organismos más complejos (Figura 5). El número de genes microARN aumentó notablemente durante la evolución temprana de los bilaterios (Grimson et al., 2008; Wheeler et al., 2009), y sus funciones se dedujeron en el último ancestro común de los bilaterios antes de la divergencia entre protostomas y deuterostomas (Christodoulou et al., 2010). Desde entonces, se han obtenido cientos de genes microARN adicionales con la evolución de tipos celulares y tejidos más especializados en organismos complejos. Se han identificado genes de microARN incluso en esponjas metazoarias primitivas, plantas y en dos especies de eucariotas unicelulares. Por lo tanto, es posible que los microARN hayan surgido varias veces durante la evolución, incluido el linaje primitivo de los animales multicelulares hace unos 600 millones de años, o que el antepasado de las plantas y los animales desarrollara microARN hace ya mil millones de años (Moran et al., 2017). En particular, el hecho de que muchos genes de microARN evolutivamente antiguos se conserven en organismos que evolucionaron posteriormente y que estos genes rara vez se pierdan a lo largo de la evolución demuestra su papel fundamental en la regulación génica.
The essential roles of microRNAs in metazoan development and for tissue function have been demonstrated through the ablation of components in the microRNA biogenesis pathway. The loss of Dicer, which processes pre-miRNAs in the cytoplasm, is embryonically lethal in mice and zebrafish (Bernstein et al., 2003; Wienholds et al., 2003). The removal of individual or groups of microRNA genes in fruit flies and mice also causes strong phenotypes (Bartel, 2018). However, the roles of individual microRNA genes can be obscured, likely due to redundant roles of several microRNA genes that share target-defining seed sequences. While the redundancy in the system represents a barrier to studying the function of single microRNA genes, it also demonstrates the robustness of the system and explains why it cannot be easily manipulated by, e.g., viruses.
To highlight the fundamental role of microRNAs, it is important to note that the most evolutionarily conserved microRNA genes, those shared among bilaterian organisms, function early in embryonic development, whereas microRNAs that evolved specifically in mammals function at later stages of embryonic development (DeVeale, Swindlehurst-Chan and Blelloch, 2021). In contrast, species-specific microRNA genes generally play roles in adult cell-types rather that in embryonic development. These patterns are evident from the systematic knockout experiments on microRNA genes of varying evolutionary conservation. The specific regulatory roles of microRNAs during animal development include developmental timing, the formation and stability of cell fates, general physiology, and homeostasis (DeVeale, Swindlehurst-Chan and Blelloch, 2021).
The functions of microRNAs in adult cells and tissues have been elucidated through selective Dicer removal in transgenic mice. Early removal of Dicer1 during B-cell maturation led to a differentiation halt at the pro-B cell stage (Koralov et al., 2008). Dicer1 ablation at embryonic day 15.5 in neurons resulted in the early postnatal death, preceded by microcephaly, reduced dendritic branch elaboration, and increased dendritic spine lengths (Davis et al., 2008). In post-mitotic cerebellar Purkinje cells, Dicer1 loss at two weeks age triggered cerebellar degeneration and ataxia onset (uncoordinated muscle movement) (Schaefer et al., 2007). Similarly, loss of Dicer1 in midbrain dopaminergic neurons led to progressive neuron loss and reduced locomotor activity (Kim et al., 2007). Severe phenotypes have been observed across several other cell types and tissues, demonstrating the critical roles of microRNAs in both developmental processes and adult cell-type functions.
The importance of microRNAs for human development and function becomes apparent through syndromes associated with mutations in specific microRNA genes or components of the biogenesis pathway. The DICER1 syndrome is a rare inherited disorder caused by a mutation in the DICER1 gene, which predisposes individuals to tumors in the kidney, thyroid, ovary, cervix, testicle, brain, eye, and lung. Often, one allele of DICER1 has a germline mutation that renders it nonfunctional, lowering the amounts of functional DICER1 protein in cells. These individuals are vulnerable for additional somatic mutations, and as a consequence, often develop tumors during childhood (Foulkes, Priest and Duchaine, 2014).
The base-pairing portion (i.e., the seed region) of individual microRNA genes is short, making them less likely to be altered by chance mutations. However, there are known mutations in the seed sequence of microRNA genes that are linked to disease. These include mutations in miRNA-96 that are associated with progressive hearing loss (Mencía et al., 2009; Soldà et al., mutations in miRNA-184 causing EDICT syndrome, a rare eye disease with iris hypoplasia, endothelial dystrophy, and congenital cataract (Hughes et al., 2011; Iliff, Riazuddin and Gottsch, 2012; Lechner et al., 2013), and mutations in miRNA-140-5p resulting in a congenital skeletal disorder (Grigelioniene et al., 2019). Advances are being made in developing microRNA-based diagnostics and therapeutics for diseases, such as metabolic disorders, cardiovascular disease, neurodegenerative conditions, and cancer.
Summary
Gracias al descubrimiento seminal de Ambros y Ruvkun, y a los numerosos colegas que se basaron en sus hallazgos, se ha revelado una nueva dimensión de la regulación génica. Mientras que las proteínas del núcleo regulan la transcripción y el empalme del ARN, los microARN controlan la traducción y degradación del ARNm en el citoplasma. Esta inesperada capa de regulación génica postranscripcional tiene una importancia crítica a lo largo del desarrollo animal y en tipos de células adultas, y es esencial para la compleja vida multicelular.
Rickard Sandberg, PhD, Professor at Karolinska Institutet, (Rickard.Sandberg@ki.se), Member of the Nobel Committee
Key references
Rosalind C. Lee, Rhonda L. Feinbaum and Victor Ambros (1993) “The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14”. Cell, 75(5), pp. 843–854.
Bruce Wightman, Ilho Ha, and Gary Ruvkun (1993) “Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans”. Cell, 75(5), pp. 855–862.
Amy E. Pasquinelli, Brenda J. Reinhart, Frank Slack, Mark Q. Martindale, Mitzi I. Kurodak, Betsy Maller, David C. Hayward, Eldon E. Ball, Bernard Degnan, Peter Müller, Jürg Spring, Ashok Srinivasan, Mark Fishman, John Finnerty, Joseph Corbo, Michael Levine, Patrick Leahy, Eric Davidson & Gary Ruvkun (2000) “Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA”. Nature, 408(6808), pp. 86–89.
*these authors contributed equally to this work.
The Nobel Assembly, consisting of 50 professors at Karolinska Institutet, awards the Nobel Prize in Physiology or Medicine. Its Nobel Committee evaluates the nominations. Since 1901 the Nobel Prize has been awarded to scientists who have made the most important discoveries for the benefit of humankind.
Nobel Prize® is the registered trademark of the Nobel Foundation
References
Ambros, V. (1989) ‘A hierarchy of regulatory gene controls a larva-to-adult developmental switch in C. elegans’, Cell 57(1), pp. 49–57. Available at: https://doi.org/10.1016/0092-8674(89)90171- 2.
Ambros, V. (2008) ‘The evolution of our thinking a bout microRNAs’, Nature Medicine, 14(10), pp. 1036–1040. Available a https://doi.org/10.1038/nm1008-1036.
Ambros, V. and Horvitz, H.R. (1984) ‘Hetero’chroni mutants of the nematode Caenorhabditis elegans’, Science (New York, N.Y. ), 226(4673), pp. 409–416. Available at: https://doi.org/10.1126/science.64948 91.
Arasu, P., Wightman, B. and Ruvkun, G. (1991) ‘Temporal regulation of lin-14 by the antagonistic action of two othe heterochronic genes, lin-4 and lin-28’, Genes & Development, 5(10), pp. 1825–1833. Available at: https://doi.org/10.1101/gad.5.10.1825.
Bartel, D.P. (2004) ‘MicroRNAs: genomics, biogenes is, mechanism, and function’, Cell, 116(2), pp. 281–297. Available a https://doi.org/10.1016/s0092-8674(04)00045-5.
Bartel, D.P. (2018) ‘Metazoan MicroRNAs’, Cell, 173(1), pp. 20–51. Available at: https://doi.org/10.1016/j.cell.2018.03.006 .
Bernstein, E. et al. (2001) ‘Role for bidentate ribonuclease in the initiation step of RNA interference’, Nature, 409(6818), pp. 363–366. Available at: https://doi.org/10.1038/35053110.
Bernstein, E. et al. (2003) ‘Dicer is essen tial for mouse development’, Nature Genetics, 35(3), pp. 215–217. Available at: https://doi.org/10.1038/ng1253.
Brennecke, J. et al. (2005) ‘Principles o microRNA-target recognition’, PLoS biology, 3(3), p. e85. Available at: https://doi.org/10.1371/journal.pbio.0030085.
Chalfie, M., Horvitz, H.R. and Sulston, J.E. (1981) ‘Mutations that lead to reiterations in the cell lineages of C. elegans’, Cell, 24(1), pp. 59–69. Available at: https://doi.org/10.1016/0092-8674(81)90501-8.
Christodoulou, F. et al. (2010) ‘Ancient animal microRNAs and the evolution of tissue identity’, Nature, 463(7284), pp. 1084–1088. Available at: https://doi.org/10.1038/nature08744.
Davis, T.H. et al. (2008) ‘Conditional Loss of Dicer Disrupts Cellular and Tissue Morphogenesis in the Cortex and Hippocampus’, The Journal of Neuroscience, 28(17), pp. 4322–4330. Available at: https://doi.org/10.1523/JNEUROSCI.4815-07.2008.
DeVeale, B., Swindlehurst-Chan, J. and Blelloch, R. (2021) ‘The roles of microRNAs in mouse development’, Nature Reviews. Genetics, 22(5), pp. 307–323. Available at: https://doi.org/10.1038/s41576-020-00309-5.
Farh, K.K.-H. et al. (2005) ‘The widespread impact of mammalian MicroRNAs on mRNA repression and evolution’, Science (New York, N.Y.), 310(5755), pp. 1817–1821. Available at: https://doi.org/10.1126/science.1121158.
Ferguson, E.L., Sternberg, P.W. and Horvitz, H.R. (1987) ‘A genetic pathway for the specification of the vulval cell lineages of Caenorhabditis elegans’, Nature, 326(6110), pp. 259–267. Available at: https://doi.org/10.1038/326259a0.
Fire, A. et al. (1998) ‘Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans’, Nature, 391(6669), pp. 806–811. Available at: https://doi.org/10.1038/35888.
Foulkes, W.D., Priest, J.R. and Duchaine, T.F. (2014) ‘DICER1: mutations, microRNAs and mechanisms’, Nature Reviews. Cancer, 14(10), pp. 662–672. Available at: https://doi.org/10.1038/nrc3802.
Grigelioniene, G. et al. (2019) ‘Gain-of-function mutation of microRNA-140 in human skeletal dysplasia’, Nature Medicine, 25(4), pp. 583–590. Available at: https://doi.org/10.1038/s41591-019-0353-2.
Grimson, A. et al. (2008) ‘Early origins and evolution of microRNAs and Piwi-interacting RNAs in animals’, Nature, 455(7217), pp. 1193–1197. Available at: https://doi.org/10.1038/nature07415.
Hamilton, A.J. and Baulcombe, D.C. (1999) ‘A species of small antisense RNA in posttranscriptional gene silencing in plants’, Science (New York, N.Y.), 286(5441), pp. 950–952. Available at: https://doi.org/10.1126/science.286.5441.950.
Horvitz, H.R. and Sulston, J.E. (1980) ‘Isolation and genetic characterization of cell-lineage mutants of the nematode Caenorhabditis elegans’, Genetics, 96(2), pp. 435–454. Available at: https://doi.org/10.1093/genetics/96.2.435.
Hughes, A.E. et al. (2011) ‘Mutation altering the miR-184 seed region causes familial keratoconus with cataract’, American Journal of Human Genetics, 89(5), pp. 628–633. Available at: https://doi.org/10.1016/j.ajhg.2011.09.014.
Iliff, B.W., Riazuddin, S.A. and Gottsch, J.D. (2012) ‘A single-base substitution in the seed region of miR-184 causes EDICT syndrome’, Investigative Ophthalmology & Visual Science, 53(1), pp. 348–353. Available at: https://doi.org/10.1167/iovs.11-8783.
Kim, J. et al. (2007) ‘A MicroRNA feedback circuit in midbrain dopamine neurons’, Science (New York, N.Y.), 317(5842), pp. 1220–1224. Available at: https://doi.org/10.1126/science.1140481.
King, N. et al. (2008) ‘The genome of the choanoflagellate Monosiga brevicollis and the origin of metazoans’, Nature, 451(7180), pp. 783–788. Available at: https://doi.org/10.1038/nature06617.
Koralov, S.B. et al. (2008) ‘Dicer ablation affects antibody diversity and cell survival in the B lymphocyte lineage’, Cell, 132(5), pp. 860–874. Available at: https://doi.org/10.1016/j.cell.2008.02.020.
Kozomara, A., Birgaoanu, M. and Griffiths-Jones, S. (2019) ‘miRBase: from microRNA sequences to function’, Nucleic Acids Research, 47(D1), pp. D155–D162. Available at: https://doi.org/10.1093/nar/gky1141.
Lagos-Quintana, M. et al. (2001) ‘Identification of novel genes coding for small expressed RNAs’, Science (New York, N.Y.), 294(5543), pp. 853–858. Available at: https://doi.org/10.1126/science.1064921.
Lau, N.C. et al. (2001) ‘An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans’, Science (New York, N.Y.), 294(5543), pp. 858–862. Available at: https://doi.org/10.1126/science.1065062.
Lechner, J. et al. (2013) ‘Mutational analysis of MIR184 in sporadic keratoconus and myopia’, Investigative Ophthalmology & Visual Science, 54(8), pp. 5266–5272. Available at: https://doi.org/10.1167/iovs.13-12035.
Lee, R.C. and Ambros, V. (2001) ‘An extensive class of small RNAs in Caenorhabditis elegans’, Science (New York, N.Y.), 294(5543), pp. 862–864. Available at: https://doi.org/10.1126/science.1065329.
Lee, R.C., Feinbaum, R.L. and Ambros, V. (1993) ‘The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14’, Cell, 75(5), pp. 843–854. Available at: https://doi.org/10.1016/0092-8674(93)90529-y.
Lewis, B.P. et al. (2003) ‘Prediction of mammalian microRNA targets’, Cell, 115(7), pp. 787–798. Available at: https://doi.org/10.1016/s0092-8674(03)01018-3.
Lewis, B.P., Burge, C.B. and Bartel, D.P. (2005) ‘Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets’, Cell, 120(1), pp. 15–20. Available at: https://doi.org/10.1016/j.cell.2004.12.035.
Mencía, A. et al. (2009) ‘Mutations in the seed region of human miR-96 are responsible for nonsyndromic progressive hearing loss’, Nature Genetics, 41(5), pp. 609–613. Available at: https://doi.org/10.1038/ng.355.
Moran, Y. et al. (2017) ‘The evolutionary origin of plant and animal microRNAs’, Nature Ecology & Evolution, 1(3), pp. 1–8. Available at: https://doi.org/10.1038/s41559-016-0027.
Pasquinelli, A.E. et al. (2000) ‘Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA’, Nature, 408(6808), pp. 86–89. Available at: https://doi.org/10.1038/35040556.
Pfeffer, S. et al. (2004) ‘Identification of virus-encoded microRNAs’, Science (New York, N.Y.), 304(5671), pp. 734–736. Available at: https://doi.org/10.1126/science.1096781.
Reinhart, B.J. et al. (2000) ‘The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans’, Nature, 403(6772), pp. 901–906. Available at: https://doi.org/10.1038/35002607.
Ruvkun, G. et al. (1989) ‘Molecular genetics of the Caenorhabditis elegans heterochronic gene lin-14’, Genetics, 121(3), pp. 501–516. Available at: https://doi.org/10.1093/genetics/121.3.501.
Ruvkun, G. and Giusto, J. (1989) ‘The Caenorhabditis elegans heterochronic gene lin-14 encodes a nuclear protein that forms a temporal developmental switch’, Nature, 338(6213), pp. 313–319. Available at: https://doi.org/10.1038/338313a0.
Ruvkun, G., Wightman, B. and Ha, I. (2004) ‘The 20 years it took to recognize the importance of tiny RNAs’, Cell, 116(2 Suppl), pp. S93-96, 2 p following S96. Available at: https://doi.org/10.1016/s0092-8674(04)00034-0.
Schaefer, A. et al. (2007) ‘Cerebellar neurodegeneration in the absence of microRNAs’, Journal of Experimental Medicine, 204(7), pp. 1553–1558. Available at: https://doi.org/10.1084/jem.20070823.
Schwarz, D.S. et al. (2003) ‘Asymmetry in the assembly of the RNAi enzyme complex’, Cell, 115(2), pp. 199–208. Available at: https://doi.org/10.1016/s0092-8674(03)00759-1.
Soldà, G. et al. (2012) ‘A novel mutation within the MIR96 gene causes non-syndromic inherited hearing loss in an Italian family by altering pre-miRNA processing’, Human Molecular Genetics, 21(3), pp. 577–585. Available at: https://doi.org/10.1093/hmg/ddr493.
Stark, A. et al. (2003) ‘Identification of Drosophila MicroRNA targets’, PLoS biology, 1(3), p. E60. Available at: https://doi.org/10.1371/journal.pbio.0000060.
Stark, A. et al. (2005) ‘Animal MicroRNAs confer robustness to gene expression and have a significant impact on 3’UTR evolution’, Cell, 123(6), pp. 1133–1146. Available at: https://doi.org/10.1016/j.cell.2005.11.023.
Wheeler, B.M. et al. (2009) ‘The deep evolution of metazoan microRNAs’, Evolution & Development, 11(1), pp. 50–68. Available at: https://doi.org/10.1111/j.1525-142X.2008.00302.x.
Wienholds, E. et al. (2003) ‘The microRNA-producing enzyme Dicer1 is essential for zebrafish development’, Nature Genetics, 35(3), pp. 217–218. Available at: https://doi.org/10.1038/ng1251.
Wightman, B. et al. (1991) ‘Negative regulatory sequences in the lin-14 3’-untranslated region are necessary to generate a temporal switch during Caenorhabditis elegans development’, Genes & Development, 5(10), pp. 1813–1824. Available at: https://doi.org/10.1101/gad.5.10.1813.
Wightman, B., Ha, I. and Ruvkun, G. (1993) ‘Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans’, Cell, 75(5), pp. 855–862. Available at: https://doi.org/10.1016/0092-8674(93)90530-4.
https://www.nobelprize.org/prizes/medicine/2024/advanced-information/
La Biología es al siglo XXI lo que la Física fue al siglo XX.
Su papel es fundamental en procesos como la diferenciación de las células y su alteración puede influir en enfermedades como el cáncer.
Sebastián Chávez de Diego
Catedrático de la Universidad de Sevilla y miembro del grupo de Expresión Génica en Eucariontes
La concesión del Nobel de Medicina 2024 a Victor Ambros y Gary Ruvkun es un gran acierto de la Academia sueca. Las contribuciones de Ambros y Ruvkun supusieron un cambio de paradigma en nuestra visión de cómo se controla la información contenida en el genoma, añadiendo una nueva dimensión. A partir de ese momento se reveló que los genes no solo se regulaban encendiéndose o apagándose en los cromosomas, sino que su expresión se controlaba también modificando la estabilidad y efectos de sus productos inmediatos (los ARN mensajeros).
Su identificación de pequeñas moléculas de ARN como reguladores postranscripcionales revolucionó nuestro conocimiento del desarrollo corporal de los organismos pluricelulares complejos, como el ser humano, y supuso un elemento imprescindible para entender el comportamiento celular en situaciones patológicas como el cáncer.
Guillermo Peris Ripollés
Profesor titular de la Universitat Jaume I y bioinformático del grupo Elementos Transponibles en desarrollo y enfermedad dirigido por la doctora Sara H. Heras, en el centro de Genómica y oncología GENYO de Granada
Este año el premio Nobel de Medicina se ha otorgado a Victor Ambros y Gary Ruvkun por el descubrimiento de los microARN. Estas secuencias ARN juegan un papel fundamental en la expresión génica, es decir, en qué genes o secuencias de ADN se transcriben en ARN y traducen a proteína en cada célula, dando una función concreta a cada tipo celular. Se denomina regulación post-transcripcional ya que, al contrario que otros mecanismos epigenéticos que también regulan la expresión génica, como la metilación del ADN, se produce después de que se ha transcrito de ADN a ARN.
La denominación de microARN proviene de que son unas secuencias muy pequeñas, de tan solo 22 nucleótidos, pero su potencial en la célula es enorme. De hecho, aumentos o disminuciones de su expresión están asociados a distintas enfermedades. Me hace una particular ilusión este premio ya que en nuestro de grupo de investigación estudiamos cómo afecta la desregulación de los microARN a diversas enfermedades, como por ejemplo tumores, y más en concreto a una enfermedad rara, el síndrome 22q11, en el que se pierde un gen muy importante para la fabricación de estas secuencias.
Sònia Guil
Líder del Grupo de Regulación del ARN y Cromatina del Instituto de Investigación contra la Leucemia Josep Carreras
Ha sido una gran alegría conocer los galardonados de este año con el Nobel de Fisiología o Medicina. Los doctores Ambros y Ruvkun revolucionaron nuestra comprensión de los programas celulares que determinan la identidad de nuestras células, descubriendo que la expresión de los genes se determina no solo en el núcleo sino también en el citoplasma celular, cuando se convierten las instrucciones del ARN en proteína. Lo crucial de sus descubrimientos es que la molécula que controla este paso es otro ARN, de muy pequeño tamaño, que pertenece a un tipo de moléculas casi desconocidas hasta el momento: los ARNs no codificantes. Estos hallazgos abrieron todo un campo importantísimo en la biología molecular, ya que estos pequeños ARNs se están usando décadas después como herramientas terapéuticas para controlar genes o como marcadores de enfermedades en la práctica clínica.
Y destaco especialmente que el descubrimiento de estos pequeños ARNs tuvo lugar en unos gusanos diminutos (1mm) de nombre complejo (Caenorhabditis elegans) y usados en investigación básica, aunque ya pocos años después se vio que este nuevo mecanismo celular está conservado evolutivamente y es de importancia clave en humanos también. Por tanto, reconocer el trabajo de estos investigadores es poner de relieve lo esencial de la investigación básica (incluyendo organismos modelos no humanos), lo cual muchas veces es poco valorado por las decisiones en política científica.
Gemma Marfany
Catedrática de Genética de la Universitat de Barcelona (UB) y jefa de grupo del CIBERER
Me parece muy interesante este Premio Nobel ya que premia a investigadores que realizaron investigación básica que luego se ha demostrado que es muy importante para comprender la regulación de la expresión de los genes. Habitualmente, explicamos que los genes son una secuencia de ADN que codifica para una proteína concreta, y que para que se pueda decodificar la información genética, el ADN se transcribe a ARN para ser luego traducido a proteínas. Sin embargo, y aunque todos los genes se transcriben, no todos codifican para proteínas. Hay genes para los que el ARN no es traducido, sino que el ARN funciona como tal.
Los ganadores del Premio Nobel de este año, Ambros y Ruvkun, justamente descubrieron un mecanismo de regulación genética que se basa en el uso de ARNs muy pequeños, de ahí el nombre de microARNs, cuya función es unirse a los ARNs de otros genes para silenciarlos y que no produzcan proteína. Es decir, que hacen la función inversa, impedir y bloquear que se traduzcan otros genes. Ambros y Ruvkun descubrieron estos genes en el nemátodo C.elegans y al principio no se les hizo mucho caso porque se pensó que era un mecanismo excepcional que solo debía actuar en organismos muy concretos, hasta que se ha descubierto que es un mecanismo de regulación muy efectivo y universal en los organismos pluricelulares. Aunque los genes de microARN son muy pequeños (de 21 a 25 nucleótidos), existen enfermedades genéticas hereditarias causadas por mutaciones en estos genes y, como consecuencia, se desregulan muchos genes distintos, pudiendo alterar varias vías de señalización o metabólicas y afectar a distintos órganos.
Mª del Carmen de Andrés González
Investigadora Miguel Servet estabilizada del Servicio Gallego de Salud, Instituto de Investigación Biomédica de A Coruña (INIBIC)
Afirmo sin temor a equivocarme que hoy todos los científicos que trabajamos en el campo de la epigenética estamos tremendamente contentos tras saber que el Premio Nobel de Medicina o Fisiología 2024 ha recaído en esas fascinantes pequeñas moléculas de ARN no codificante: los microARNs.
Pocas moléculas han sido implicadas en tantos procesos fisiológicos vitales como los microRNAs, mediante la represión de sus genes diana están involucrados en procesos inmunológicos, cancerígenos, del desarrollo o en procesos inflamatorios, expresándose a su vez en una gran variedad de organismos.
Es un premio muy merecido a los investigadores que descubrieron el primer microARN, demostrando una vez más que la ciencia básica es el primer paso para llegar a la traslación clínica.
Fátima Gebauer Hernández
Investigadora especializada en los mecanismos de regulación de la traducción del ARNm y el cáncer
El premio Nobel de Medicina o Fisiología recae este año en Victor Ambros y Gary Ruvkun, por el descubrimiento del microARN y su papel en la regulación postranscripcional de los genes. Los microARNs, como su nombre indica, son pequeñas moléculas de ARN de 20-22 nucleótidos que se unen por complementariedad de bases a moléculas de ARN más grandes, como los ARN mensajeros (ARNm) que codifican cada una de nuestras proteínas. Al unirse, los microARNs regulan la estabilidad y la eficiencia de traducción del ARNm, condicionando los niveles de proteínas en la célula y, en consecuencia, la fisiología celular. La regulación por microARNs es muy importante para mantener una fisiología sana, y fallos en esta regulación pueden ocasionar no solo problemas durante el desarrollo embrionario, sino también contribuir a enfermedades como el cáncer.
Me alegro mucho de que el premio Nobel de este año haya recaído en el descubrimiento de los microARNs por dos motivos. Primero, porque se descubrieron en el gusano C. elegans, y es un ejemplo más de cómo la cienca básica es tan fundamental para el avance de la Medicina, y de por qué los gobiernos deben financiar la ciencia básica. Segundo, porque junto con otros Premios Nobel anteriores, como el del año pasado a Katalin Karikó y Drew Weissman por las modificaciones del ARNm que fueron esenciales para el desarrollo de vacunas contra la pandemia COVID-19, resaltan la relevancia del ARN y la regulación post-transcripcional. ¡Son buenos tiempos para el ARN!
Mª Esperanza Cerdán
Catedrática de Bioquímica y Biología Molecular en el departamento de Biología de la Universidad de A Coruña
Aunque todas las células de nuestro organismo contienen básicamente la misma información genética, en no todas llega a expresarse de la misma forma, y este proceso que llamamos ‘regulación de la expresión génica’ es lo que permite que unas células sean distintas unas de otras. No expresa los mismos genes una célula epitelial o una neurona, por ejemplo. Tampoco expresa los mismos genes una célula sana que una cancerosa. Hay muchos procesos que suceden en la célula que contribuyen a hacer posible estas diferencias y en ellos intervienen interacciones entre moléculas.
El interés de las investigaciones de los científicos galardonados consiste en haber puesto en evidencia la existencia de un nuevo mecanismo, hasta entonces desconocido, que es la participación de moléculas muy pequeñas de ARN (microARNs) que controlan el proceso mediante mecanismos que suceden una vez que los ARNs mensajeros, copia de la información de algunos genes, ya han sido sintetizados. El reconocimiento entre el AR mensajero y el microARN es muy sencillo, simplemente porque sus bases son complementarias, pero eso dirige a una serie de proteínas hacia ese mensajero, determinando que pueda ser degradado y su información no sea expresada en la célula.
Los microARNs son importantes herramientas en el diagnóstico de determinados tipos de cáncer y tienen la ventaja de que se pueden detectar mediante biopsia líquida, en análisis de sangre. Su descubrimiento ha permitido el desarrollo de nuevas herramientas para la investigación biológica en todos los ámbitos. También pueden ser sintetizados fácilmente lo que favorece sus posibles aplicaciones terapéuticas.
Carlos Fernández-Hernando
Catedrático Anthony N. Brady, director del Programa de Biología Vascular y Terapéutica (VBT), departamentos de Medicina Comparada y Patología, Centro de Metabolismo Molecular y Sistémico de Yale (YMSM), facultad de Medicina de la Universidad de Yale (Estados Unidos)
Me parece un premio Nobel muy merecido. Los dos galardonados descubrieron pequeñas moléculas de ARN que regulan post-transcripcionalmente la expresión génica de la gran mayoría de genes. Ruvkun y Ambros identificaron estas moléculas estudiando el desarrollo del nematodo C. elegans cuando observaron que una pequeña molécula de ARN, Lin-4, suprimía la expresión de Lin-14 durante el proceso de desarrollo de este organismo. La alteración en la expresión de estas moléculas está asociada a numerosos procesos patofisiológicos, incluidos el cáncer, las enfermedades neurodegenerativas y cardiometabólicas. Existe la posibilidad de manipular la expresión de estas moléculas para el tratamiento de estas enfermedades. Por otro lado, el descubrimiento de los microARNs supone un hallazgo muy relevante, puesto que estas moléculas controlan numerosos procesos celulares y fisiológicos básicos como el desarrollo, la proliferación celular y el metabolismo del colesterol.
Noemí Rotllan Vila
Investigadora principal, Grupo de Fisiopatología de las Enfermedades Relacionadas con Lípidos, Instituto de Investigación Biomédica Sant Pau (IIB-Sant Pau), CIBER de Diabetes y Enfermedades Metabólicas Asociadas (CIBERDEM)
Personalmente, para mí es una alegría la concesión del Premio Nobel de Fisiología o Medicina a dos grandes científicos como son Victor Ambros y Gary Ruvkun, por sus estudios que iniciaron en los 80’s como estudiantes postdoctorales, y que luego continuaron como investigadores independientes sobre la regulación génica y cómo esta permite que distintos tipos celulares se desarrollen o no en un determinado momento.
Mi investigación también se centra en el fascinante mundo de los microARNs, pequeñas secuencias de ARN no codificante de cadena simple (entre 19-22 nucleótidos) que regulan la expresión génica a nivel postranscripcional, ya sea inhibiendo la traducción o promoviendo la degradación del ARN mensajero (ARNm). Desde su descubrimiento, se ha revelado cómo un solo microARN puede regular más de 100 ARNm, y de igual manera, cómo un ARNm puede estar regulado por varios microARNs. Además, los microARNs están implicados tanto en procesos de salud como en el desarrollo de enfermedades.
Me gustaría destacar que los microARNs son dianas terapéuticas muy prometedoras y pueden utilizarse como biomarcadores para diversas enfermedades, especialmente en cáncer y enfermedades cardiovasculares. De hecho, numerosos estudios preclínicos han explorado la sobreexpresión o inhibición de determinados microARNs. Algunos de estos estudios han avanzado hasta ensayos clínicos, como es el caso de Miravirsen, el primer fármaco dirigido específicamente a un microARN, que actualmente se encuentra en fase II (evaluación de seguridad y eficacia) en pacientes. Sin embargo, debemos ser cautelosos, ya que aún queda mucho por investigar y desarrollar.
Otro aspecto que me gustaría destacar es que sus estudios se enmarcan en la investigación básica, concretamente en modelos con pequeños gusanos (C. elegans), que a menudo son subestimados. Es fundamental resaltar la importancia de estos estudios para el avance del conocimiento científico.
Alicia González-Martín
Investigadora del departamento de Bioquímica de la UAM, especialista en el papel de los microARN en la inmunología tumoral
Es un gran acierto y una alegría que el Premio Nobel de Medicina de este año haya sido otorgado a Victor Ambros y Gary Ruvkun por su descubrimiento de los microARNs y su papel en la regulación génica postranscripcional. Este hallazgo es de gran importancia, al transformar nuestra comprensión de la regulación de la expresión génica y atribuir funciones críticas a una fracción del genoma humano que anteriormente se consideraba 'ADN basura' por no codificar proteínas. Estos pequeños ARNs no codificantes desempeñan papeles fundamentales en prácticamente todos los procesos celulares, incluyendo el desarrollo, la inmunidad y la progresión de enfermedades, mediante la regulación simultánea de múltiples genes, lo que genera efectos funcionales potentes. El impacto de los microARNs en la medicina es evidente, posicionándolos como moléculas clave en el desarrollo de nuevas terapias para enfermedades complejas, como el cáncer y las enfermedades autoinmunes, entre muchas otras.
Pilar Martín Fernández
Jefa del grupo Moléculas Reguladoras de la Inflamación en el Centro Nacional de Investigaciones Cardiovasculares (CNIC)
Este premio Nobel destaca la enorme relevancia de los microARNs (miRNAs) en la regulación post-transcripcional de la expresión génica, un campo en rápido crecimiento con aplicaciones directas en la medicina de precisión, gracias a los avances en la terapia con ARN. Algunos de estos avances han sido reconocidos con premios Nobel en años anteriores. Tras el éxito de las vacunas de ARN administradas a millones de personas, se ha demostrado que la terapia con ARN es un área llena de posibilidades prometedoras. Los microARNs, estudiados desde los años 80 por Victor Ambros y Gary Ruvkun, quienes fueron becarios postdoctorales en el laboratorio de Robert Horvitz —galardonado con el Nobel en 2002 junto a Sydney Brenner y John Sulston por sus descubrimientos sobre la regulación genética del desarrollo de órganos y la muerte celular programada—, continúan revelando mecanismos fundamentales para la biología y la medicina moderna.
Los miRNAs son pequeñas moléculas de ARN no codificantes, de aproximadamente 22 nucleótidos de longitud, que regulan la expresión génica a nivel post-transcripcional. Estos miRNAs actúan uniéndose a secuencias complementarias en el ARN mensajero de genes específicos, lo que conduce su degradación o a la inhibición de su traducción, bloqueando así la producción de proteínas. Este mecanismo de silenciamiento génico es esencial para el control de procesos biológicos clave, como el desarrollo celular, la diferenciación, y la respuesta a estrés o enfermedades, y está implicado en la regulación de múltiples enfermedades, desde el cáncer hasta enfermedades cardiovasculares, las cuales representan la principal causa de mortalidad global.
Además, la identificación de perfiles específicos de miRNAs circulantes en sangre está mostrando gran potencial para la detección temprana de cánceres difíciles de diagnosticar, como el cáncer de páncreas y pulmón. Estudios recientes señalan que los miRNAs podrían utilizarse como biomarcadores en enfermedades cardiovasculares como la miocarditis, la aterosclerosis, la hipertensión o la fibrosis cardiaca.
Este reconocimiento a Victor Ambros y Gary Ruvkun refuerza la importancia de los miRNAs, abriendo puertas a aplicaciones terapéuticas y diagnósticas que podrían transformar el manejo de enfermedades tan devastadoras como el cáncer y las patologías cardiovasculares.
Conflicto de interés: Pilar Martín es inventora de una patente para el uso de nuevos microRNAs para el diagnóstico de cardiomiopatías.
Nick Stroustrup
Jefe de equipo en el Centre de Regulació Genòmica (CRG)
Victor y Gary utilizaron el asombroso poder de la genética de C. elegans para descubrir los microARN. Por aquel entonces, trabajaban para comprender cómo se desarrollan los nematodos desde embriones a adultos e identificaron genes importantes que codificaban ARN pero no proteínas, algo que no se creía que ocurriera en biología. El trabajo de Victor y Gary condujo directamente a la comprensión de que los microARN son un mecanismo crucial para la regulación de los genes en todo el árbol de la vida, incluidos los seres humanos.
Manuel Irimia
Profesor ICREA, investigador en la Universitat Pompeu Fabra (UPF), director del grupo de Transcriptómica del Desarrollo y Evolución de los Vertebrados en el Centro de Regulación Genómica (CRG) de Barcelona
El descubrimiento de los miRNA, pequeñas moléculas de RNA que modulan la cantidad de proteína producida por los genes, fue una auténtica revolución en todos los campos de la biología y medicina. En primer lugar, fueron un paso más en la deconstrucción el dogma de la biología molecular, ya que mostraba que los RNAs, no solo las proteínas podían regular la expresión de los genes del genoma. Esto abrió la puerta al descubrimiento de redes reguladoras, más y más complejas, basadas en RNAs no codificantes, incluyendo, por ejemplo, otras moléculas de RNA más largas. Segundo, a medida que iba quedando claro que la regulación por miRNAs no eran una excepción sino la regla, se estudió y demostró su relevancia en prácticamente todos los campos de la investigación biomédica: desde la evolución de los seres vivos al desarrollo embrionario y la progresión del cáncer y otras enfermedades. Los miRNAs resultaron ser una parte esencial de casi todos los circuitos génicos y, por tanto, todos los investigadores en biomedicina nos hemos topado con los miRNAs, en mayor o menor medida, en algún momento de nuestras carreras.
Este galardón es un nuevo reconocimiento a la importancia capital de la ciencia básica, a la investigación dirigida por la curiosidad. Como en muchos otros casos, unas observaciones inesperadas y potencialmente anecdóticas, acabaron derivando en una explosión de nuevas oportunidades prácticas y en una revolución en nuestro conocimiento sobre la vida.
Víctor Borrell
Profesor de Investigación del CSIC en el Instituto de Neurociencias (Alicante), donde dirige un grupo de investigación que estudia el desarrollo embrionario del cerebro y su evolución, especialista en el plegamiento de la corteza cerebral
El premio Nobel de Fisiología o Medicina de este año ha sido galardonado a dos grandes científicos, que descubrieron la existencia de los micro ARNs. Este hallazgo significó un antes y un después en el campo de la genética, puesto que puso de manifiesto un nuevo mecanismo biológico y molecular por el que las células regulan la expresión genética. La importancia clave radica en que los microARNs no son un mecanismo alternativo a lo ya conocido, sino un nivel adicional de regulación génica, que se añade y combina a todos los otros mecanismos ya conocidos. Por ello, este descubrimiento nos abrió los ojos a un nuevo mundo de posibilidades antes insospechadas, iniciando un nuevo campo de investigación que tiene y ha tenido repercusiones en muchísimas direcciones: en la investigación fundamental, a nivel de mecanismos moleculares, evolución e incluso el origen de la vida, como en la investigación aplicada al sistema de salud.
Tras su descubrimiento inicial se han identificado miles de micro ARNs diferentes, y comprendemos como los producen las células y cuáles son sus efectos. Una de las características de los miARNs es la complejidad de su forma de acción: sabemos que estas pequeñas moléculas pueden actuar solas, o en grupo, y que su forma de actuar y el efecto que producen pueden variar enormemente según el contexto: el tipo de célula o su estado. Todo ello ha cambiado para siempre nuestra forma de mirar la genética, tradicionalmente determinista.
Aunque son moléculas pequeñas, se ha descubierto que existen desde hace muchos millones de años y desempeñaron funciones clave, tanto en la evolución de nuestro cerebro, por ejemplo, como en su patología, incluyendo cánceres cerebrales infantiles que surgen en el mismo embrión.
Por todo esto, se trata de un premio más que merecido, y que pone en valor la ciencia que ‘solo’ hace avanzar el conocimiento, pero aparentemente sin utilidad social inmediata, en un mundo que exige rentabilidad a corto plazo. Hoy el comité Nobel premia un descubrimiento de Victor Ambros y Gary Ruvkun que cambió para siempre los libros de texto.
Manel Esteller
Profesor de Investigación ICREA en el Instituto contra la Leucemia Josep Carreras
Los doctores Victor Ambros y Gary Ruvkun han recibido el Premio Nobel de Fisiología o Medicina en la presente edición por el descubrimiento de los microARNs. El dogma central de la biología molecular dice que a partir de los genes (ADN) se produce una molécula llamada ARN mensajero (se parece mucho al ADN pero no es de doble cadena y en vez de “T” en su secuencia tiene “U”), que es la responsable de llevar la información a las fábricas de las células (ribosomas) que producen las proteínas (hemoglobina, insulina, etc.).
Este dogma presenta varias grietas y los investigadores mencionados descubrieron uno más: existen genes (alrededor de 1.000 en el genoma humano) que no producen ARN mensajero sino un ARN más pequeño (de 21 a 25 piezas) denominado microARN que no origina proteínas. Estos microRNAs son los reguladores de los ARN mensajeros: se unen a los mismos por complementariedad química e inhiben su función. Este fenómeno contribuye a que, aunque todas las células del cuerpo humano tienen la misma secuencia de ADN, puedan producir cantidades de proteína distinta: por ejemplo, en la retina requieres expresión elevada de rodopsina para ver, mientras que la piel expresa niveles altos de queratina.
Los hallazgos de los premiados fueron inicialmente descritos en un gusano usado ampliamente en los laboratorios, el Caenorhabditis elegans, en 1993; pero con posterioridad en el año 2000 también demostraron que sucedían en humanos. Hoy en día sabemos que los patrones de expresión de los microARNs están alterados en muchas patologías como el cáncer y sus genes están mutados en algunas enfermedades minoritarias. Existe también una investigación farmacológica muy activa para encontrar fármacos que actúen a nivel de los microARNs. ¡Es un reconocimiento bien merecido!
Alejandro Piñeiro Ugalde
Investigador Ramón y Cajal en el departamento de Bioquímica y Biología Molecular de la Universidad de Oviedo
El premio Nobel de Medicina de este año ha galardonado a los dos científicos americanos descubridores de la regulación de la expresión génica mediada por microARNs: los profesores Victor Ambros y Gary Ruvkun. Aunque todas las células de nuestro cuerpo poseen el mismo genoma (los mismos genes), no todos los genes están activos en todas ellas. Una de las claves necesarias para entender la salud y la enfermedad es la comprensión de los mecanismos que gobiernan qué genes y en qué grado deben estar activos en un momento dado en cada célula, lo que se conoce como la expresión génica. En este sentido, el trabajo de los premiados estudiando elementos reguladores del desarrollo de un pequeño gusano de laboratorio inició el descubrimiento de un amplio mecanismo de regulación de los seres vivos hasta entonces desconocido.
Estos investigadores descubrieron que los genomas también producen moléculas de ARN de muy pequeño tamaño (microARNs) que regulan los niveles de expresión de la mayor parte de los genes mediante su unión a secuencias específicas presentes en los ARNs mensajeros. Desde su descubrimiento en 1993, se han identificado más de 1.000 genes humanos de microARNs, y el trabajo de multitud de laboratorios ha revelado que estas moléculas están implicadas en la regulación de prácticamente cualquier aspecto de nuestro organismo, incluyendo el envejecimiento como ha demostrado nuestro laboratorio.
Nuria Flames
Investigadora principal del grupo de Neurobiología del Desarrollo del Instituto de Biomedicina de Valencia (CSIC)
Santiago Vernia
Investigador principal del grupo de Metabolismo y Regulación de la expresión génica del Instituto de Biomedicina de Valencia (CSIC)
Este premio a los profesores Victor Ambros y Gary Ruvkun era esperado desde hace tiempo. Sus trabajos han sido claves en el descubrimiento de los microARNs, unas moléculas que tienen una función clave en la regulación de la actividad de muchos de nuestros genes. Además, estos descubrimientos dieron paso al uso de los microARNs como marcadores de enfermedades y nuevas dianas terapéuticas —el doctor Santiago Vernia y la doctora Susana Rodriguez-Navarro, trabajan en el campo del RNA en el Instituto de Biomedicina de Valencia del CSIC (IBV-CSIC)—.
Cabe destacar que estos descubrimientos fueron en buena parte posibles gracias a la elección del organismo modelo Caenorhabditis elegans para sus investigaciones. C.elegans es un pequeño (1mm) nematodo fácil de crecer y manipular en el laboratorio pero que, a su vez, comparte con los humanos los principales tejidos y sus mecanismos de regulación, constituyendo una herramienta muy potente para caracterizar procesos fundamentales del funcionamiento de nuestras células —la doctora Nuria Flames y el doctor José Pérez son ambos especialistas en este pequeño organismo modelo—. Estrategias similares ya dieron lugar a otros premios Nobel como Mello & Fire (2006) que también utilizaron C.elegans para caracterizar otros procesos como el de interferencia de ARN, que ha dado lugar a fármacos que ya han llegado a la clínica.
- https://sciencemediacentre.es/nobel-de-medicina-o-fisiologia-para-ambros-y-ruvkun-por-el-descubrimiento-de-los-microarns-y-su
2023
https://notistecnicas.blogspot.com/2023/10/premio-nobel-de-medicina-para-los.html

No hay comentarios:
Publicar un comentario